Sopa de letrinhas do DNA
Por Bianca Leal
A-T, G-C. Do Ensino Médio à Pós-Graduação nos acostumamos com essas bases nitrogenadas: Adenina, Timina, Guanina e Citosina. Elas compõem o código do nosso DNA, que é lido e traduzido em proteínas que permitem a vida dos organismos vivos. Mas como sabemos em que ordem essas bases se colocam nos genomas?
Isso é tarefa para o sequenciamento de DNA!
PRIMEIRO, VAMOS ÀS INTRODUÇÕES
O sequenciamento corresponde à determinação da ordem das bases nitrogenadas em uma molécula de DNA, seja ela pequena ou grande. As tecnologias de sequenciamento fazem isso por metodologias variadas: nucleotídeos fluorescentes, diferença de potencial gerada por cada base, geração de luz na adição dos nucleotídeos.
Cada uma das gerações de sequenciadores apresenta características particulares quanto ao rendimento, velocidade e custo de realização, aspectos que são sempre considerados quando é necessário realizar o sequenciamento de determinada amostra.
DE GERAÇÃO EM GERAÇÃO
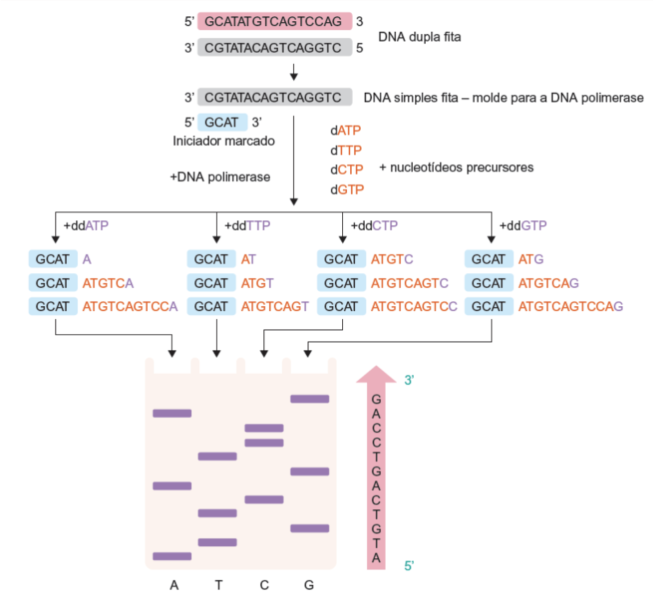
A primeira geração de sequenciadores é encabeçada pelo método de Sanger (1977), que inicialmente consistia na adição de didesoxinucleotídeos (ddNTPs) radiomarcados, que atuava como um terminador da sequência, junto ao pool de nucleotídeos (dNTPs) utilizados para amplificar o DNA. Assim seriam obtidos fragmentos de tamanhos variados terminados com bases conhecidas. Esse material era submetido a eletroforese em gel e revelado por autorradiografia, o que permitia a reconstituição da ordem dos pares de base. Esse método foi automatizado trocando a radiomarcação por nucleotídeos marcados com fluoróforos e a eletroforese em gel por eletroforese capilar.
Embora tenha revolucionado a biologia molecular e fomentado a bioinformática, sendo a grande responsável pelo Projeto Genoma Humano, a tecnologia de Sanger tinha algumas limitações. Entre elas, seu nível de sensibilidade limitado para certas aplicações.
A segunda geração de sequenciadores (next generation sequencing, NGS) começou a ser desenvolvida paralelamente ao sequenciamento de Sanger, visando à redução de custos, ao aumento do rendimento e à redução do tempo de processamento. Dois de seus mais relevantes representantes são a Illumina, marcada pelo sequenciamento massivo paralelizado, e o IonTorrent, baseado na mudança de potencial elétrico com o aumento do pH ao longo da adição de bases. Assim como no sequenciamento de Sanger, a tecnologia da Illumina se encaixa como sequenciamento por síntese.
Por fim, a terceira geração de sequenciadores pode ser definida a partir das tecnologias capazes de sequenciar moléculas únicas (single molecule sequencing, SMS) em tempo real, embora a distinção entre NGS e a terceira geração seja tema de debate. Entre as tecnologias que encabeçam esta geração estão a PacBio, com a detecção dos sinais luminosos da incorporação de bases em tempo real, sendo mais uma tecnologia de sequenciamento por síntese, e o MinION, fundamentada na mudança de potencial característica de um grupo de bases nitrogenadas ao passar pelo nanoporo.
É interessante mencionar que esta última tecnologia, fundamentada no sequenciamento por nanoporo, ganhou bastante destaque no cenário da COVID-19. Inclusive o genoma do primeiro SARS-CoV-2 circulante na América Latina foi sequenciado no Brasil pela equipe das Doutoras Ester Sabino e Jaqueline Goes utilizando o MinION.
MAS PARA QUE SEQUENCIAR O DNA?
O conhecimento do código da vida tem muitas aplicações (não é à toa que houve tanta dedicação para o Projeto Genoma Humano!).
Ter em mãos a sequência que compõe o DNA das variadas espécies permite aos pesquisadores: 1) comparar espécies diferentes, novas ou extintas a fim de estabelecer relações filogenéticas; 2) identificar genes e regiões de regulação de interesse; 3) identificar mutações em pacientes ou linhagens, que podem ou não ser importantes; 4) identificar pessoas para fins forenses.
Assim, essas tecnologias e as que estão por vir, abrem portas para aplicações desde a biologia evolutiva até as ciências biomédicas e forenses, dando ainda suporte à forte e crescente era das ômicas.
Referência
HEATHER, J. M.; CHAIN, B. The sequence of sequencers: The history of sequencing DNA. Genomics, v. 107, p. 1-8, 2016.
MENCK, C. F. Genética Molecular Básica: dos genes aos genomas, 1ª ed. Rio de Janeiro – RJ: Gen, 2017.
NBIC+. Oxford Nanopore’s MinION Device Enables Real-time COVID-19 Genome Sequencing. Disponível em: https://statnano.com/news/67579/Oxford-Nanopore%E2%80%99s-MinION-Device-Enables-Real-time-COVID-19-Genome-Sequencing. Acesso em: 08 out. 2023.
VARSOMICS. Sequenciamento Sanger: história, prática e impacto do método. Disponível em: https://blog.varsomics.com/sequenciamento-sanger-historia-pratica-e-impacto-do-metodo/. Acesso em: 08 out. 2023.
VARSOMICS. Tipos de Sequenciamento: Clássico ao NGS. Disponível em: https://blog.varsomics.com/tipos-de-sequenciamento/. Acesso em: 08 out. 2023.